从细胞最基本的各种功能原件开始,精确认识其动态工作机理,是认识生命、有效干预生命过程具备基本意义的第一步。伴随冷冻电镜技术的突破,蛋白质静态晶体结构目前能够高效获取,为突破生命科学认知局限提供了巨大便利。之后,解析蛋白质分子内部复杂部件的动态反应机理,是生命科学未来亟需突破的难题。其中,DNA/RNA聚合酶等马达分子精确动态工作机理的明晰,将为高效研发控制病毒复制的有效药物提供可行前提。遗憾的是,目前模糊状态的工作机理,使得控制病毒的有效药物研发耗时长、投入大、效率低下。
中国科学院物理研究所/北京凝聚态物理国家研究中心软物质物理实验室SM1组的谢平研究员运用广义第一性原理进行理论计算和模拟,对生命活动的核心部件——各种分子马达的工作机理进行了系统地研究。鉴于生物科学研究手段限制:传统生化实验笼统平均化、晶体结构的数据静态化和新生代单分子实验数据的分散差异性及可观测数据局限性,聚合酶分子马达等功能蛋白分子的精确动态工作机制研究举步维艰,至今不甚明了,只能给出卡通画式简单模型加以定性描述。2013年,谢平研究员提出了DNA聚合酶Klenow片段(被广泛研究的高保真聚合酶模型分子)连续动态工作机理的理论模型。该模型统一解释了当时所有传统生化和单分子技术关于这一马达分子的实验数据,并对国际同行单分子实验结果实现了高度拟合。基于此模型,谢平研究员提出Klenow聚合酶马达分子在受到外力时催化速率精确变化的理论预言。
最近,软物质物理实验室SM1组的刘玉如副研究员和李伟副研究员,运用单分子操控技术对该理论预言进行了检测,实验结果与理论预言完全吻合。研究团队自主设计组装的高通量、高时空分辨率、高计算处理能力单分子磁镊仪器操纵系统,使得纳米尺度实时高效测定Klenow聚合酶这一低持续性、多停顿的单分子催化反应速率成为可能。运用物理逻辑推理和理论计算,加上高质量实验结果的高通量分析,解析验证了DNA聚合酶Klenow在外力诱导下的催化活性变化,在实验中精确检测分子马达实时动态合成反应的速率变化。实验发现,在小外力(3.8pN)阻滞下,Klenow聚合酶的合成速率达到峰值,这一有趣的反直觉现象反映了高保真DNA聚合酶Klenow分子内部各部件之间的作用机制。
本研究首次诠释了DNA聚合酶Klenow的连续动态自动化工作机理。从DNA聚合酶分子内部原子与DNA之间相互作用隧道和关键位点的理论计算和逻辑推理,得出酶分子在催化位点处(nth position)保持最大相对结合能,从而使得酶分子在反应过程中实现于动态微扰中始终落入起始位点的化学机械偶联机理。这一工作今后在新实验数据基础上继续深化和细化,将为未来高效研发控制病毒、细菌和癌症等重大疾病的有效药物奠定前驱基础。
相关研究结果已经发表在CPL上, 并被选为编辑推荐的论文(Editor’s Suggestion)。该工作得到了国家自然科学基金委, 科技部和中国科学院的支持。
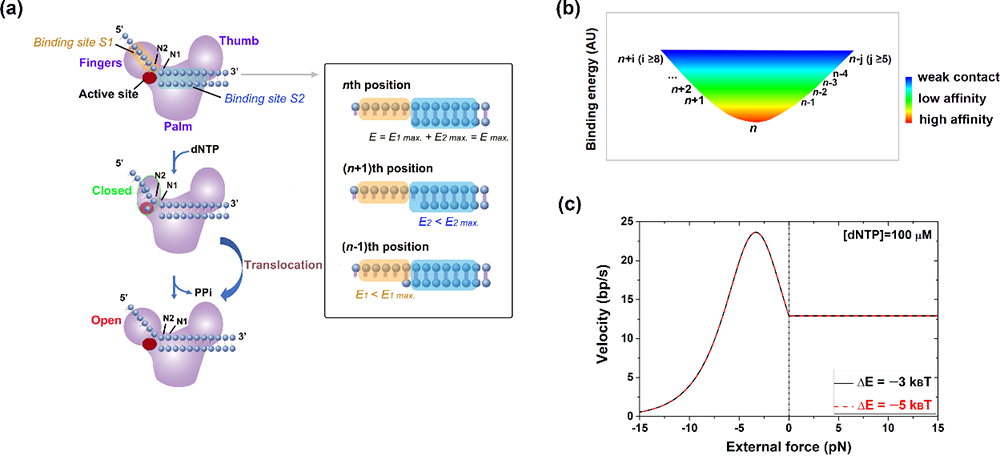
图1. DNA聚合酶(Klenow聚合酶)的自动移位机理图(a),与底物DNA不同结合位点的相对结合能(b),理论预言聚合反应在不同外力下的催化速率(c)。对DNA聚合酶分子内部原子与DNA之间相互作用隧道和关键位点的理论计算和逻辑推理,得出酶分子在催化位点处(nth position)保持最大相对结合能,从而使得酶分子在反应过程中实现于动态微扰中始终落入起始位点的化学机械偶联机理。根据酶分子内部fingers结构域不断开合和与DNA模板相互作用,提出理论预言——外力对Klenow聚合酶的催化速率具有显著影响,如(c)所示,正向外力对催化速率没有影响;反向外力在小的力值(3.8pN)左右,使催化速率显著升高,更大的反向外力使催化速率降低。
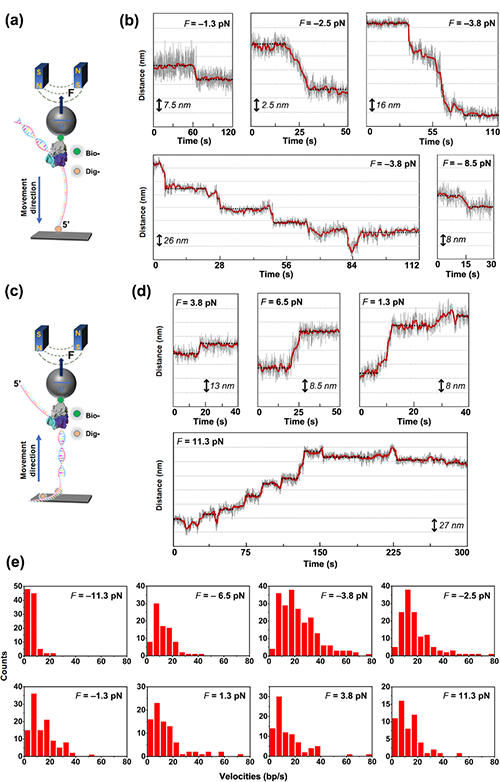
图2. 单分子磁镊技术对DNA聚合酶的催化反应进行实时动态监测。(a)和(c)分别为监测反向和正向外力的实验装置示意图。(b)和(d)分别为反向和正向外力作用下酶催化反应的动态曲线。(e)为不同外力作用下的酶催化速率分布统计。
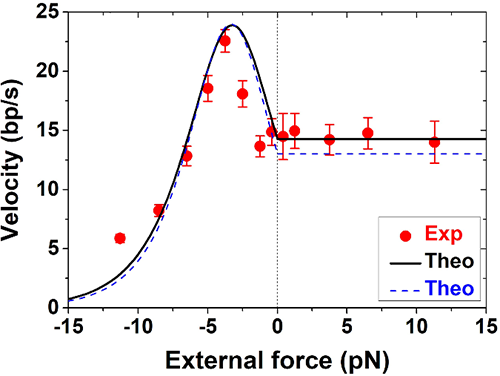
图3. 理论预言结果与实验测量结果吻合。实验测量结果为红色圆点表示;运用本研究实验体系微调后的参数拟合理论结果显示为黑色实线;运用历史文献参数拟合的理论结果显示为蓝色虚线。

